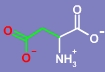
Os oxigênios das hidroxilas do grupo carboxílico da cadeia lateral estão coloridos em vermelho.
Introdução
Muitas proteínas atuam como enzimas, ou seja, como catalisadores
biológicos de reações químicas
específicas.
As proteases são enzimas que catalisam a hidrólise
(quebra
de ligação covalente com participação de
uma
molécula de água) das ligações
peptídicas
de outras proteínas, clivando-as em fragmentos de
aminoácidos
menores.
Essas enzimas atuam em diversos processos fisiológicos e
celulares.
Por exemplo, quando comemos carne, as proteínas que a
compõem
são clivadas para que os aminoácidos sejam absorvidos por
proteases específicas.
Neste capítulo, vamos examinar em detalhes a estrutura de uma
protease
para entender como a posição de aminoácidos
específicos
é essencial para promover a catálise da quebra de uma
ligação
peptídica.
Dos
inumeráveis
papéis fisiológicos/celulares de proteases, um importante
papel é na maturação de outras proteínas.
Muitos vírus,
ao invés de expressarem suas proteínas
separadamente,
expressam uma cadeia polipeptídica única composta de
diversas
proteínas diferentes unidas. Depois, uma protease também
expressa pelo vírus cliva a cadeia única em pontos
específicos,
liberando as diversas proteínas.
| ver
animação da função das proteases virais |
Este é
o caso da protease de HIV-1. Como esta enzima é fundamental para
que o vírus se replique, a inibição dela poderia
ser útil
no controle da doença. Atualmente são conhecidas
diferentes
moléculas capazes de inibir esta protease, como as drogas
adenovir e saquinavir.
Vamos estudar
a estrutura da retropepsina de HIV-1 e entender como ela executa sua
atividade proteolítica e como se dá a
inibição
da atividade de protease.
| carregar modelo
atômico |
A retropepsina
de HIV-1 funcional é um homodímero.
| colorir monômeros separadamente |
Gire a
molécula
para verificar a simetria da estrutura formada.
Vamos
visualizar
somente o esqueleto carbônico para verificar mais facilmente a
simetria
do dímero.
| ver esqueleto carbônico |
Repare
também
como as pontes de hidrogênio entre os aminoácidos da
proteína
contribuem enormemente para manutenção da estrutura.
| ver pontes de hidrogênio (em amarelo) |
Vamos visualizar
novamente a proteína com os raios atômicos.
| ver raios atômicos |
Rotacione o modelo
da retropepsina em estudo e verifique a existência de uma
cavidade
vazia na proteína.
| selecionar
aminoácidos para indicar localização da abertura
(em vermelho) |
Este espaço
vazio é ocupado pelo substrato (proteína a ser clivada)
da
retropepsina de HIV-1. Dentro da cavidade, localizam-se
3 aminoácidos de cada monômero que são
responsáveis
pela clivagem do substrato. O sítio catalítico é
então
uma região da proteína que forma um compartimento onde a
catálise de uma certa reação química
é mais
favorável. No caso das proteases, esta reação
é
a hidrólise da ligação peptídica.
| mostrar aminoácidos catalíticos da retropepsina (nos dois monômeros em verde) |
Gire a molécula
para ver uma região colorida em verde,
dentro da cavidade que abriga o substrato.
Para
visualizar
melhor o sítio catalítico, vamos apagar uma das cadeias.
| apagar cadeia B (visualizar apenas esqueleto peptídico) |
Os aminoácidos
coloridos em verde
são aqueles diretamente responsáveis pela catálise
da hidrólise (Asp25, Thr26 e Gly27). Apesar da catálise
requerer
que toda a proteína esteja intacta, sabe-se que certas
mutações
na estrutura não causam um grande impacto no funcionamento da
protease.
Entretanto, mutações em um dos 3 aminoácidos
Asp25,
Thr26 e Gly27 causam. Por isso estes são considerados os
aminoácidos
associados diretamente à catálise.
Veja que na
cadeia B, representada apenas pelo esqueleto peptídico,
também
há aminoácidos coloridos em verde, já que a
proteína
é dimérica.
Assim, o
sítio
catalítico desta protease só existe quando a
proteína
está em forma dimérica.
Vamos ver a
cavidade
que abriga o substrato de outra forma.
| preencher cadeia B |
Para ver
diretamente
os aminoácidos catalíticos, vamos "remover" os
aminoácidos
que fecham o sítio catalítico. Na verdade, é
justamente
isso que ocorre, há uma aba ("flap") que forma uma tampa da
cavidade
onde o substrato se localiza, sendo que ela pode se abrir e fechar,
permitindo
a entrada de novos substratos e mantendo-os dentro do sítio
ativo
para faciliar a catálise da hidrólise da
ligação
peptídica.
| ver "flap" como esqueleto de aminoácidos |
Agora é
possível observar diretamente o sítio catalítico,
através do "flap".
Gire a
molécula
e perceba que o "flap" forma uma aba que simplesmente se estende por
sobre
o sítio ativo.
| remover flap |
| recolocar flap |
Rotacionando a molécula, pode-se perceber que o sítio catalítico encontra-se enterrado no meio da proteína.
Tendo analisado
a localização do sítio catalítco desta
enzima,
vamos estudar como a posição de aminoácidos
específicos
está relacionada à catálise enzimática.
Dos 6
aminoácidos
(3 de cada monômero) que constituem o sítio ativo desta
protease,
os aspartatos são aqueles que de fato catalisam a
reação.
O aspartato é um aminoácido que possui como cadeia
lateral,
um grupo carboxílico.
| ver Asp25 das duas cadeias |
Embora não
se conheça o mecanismo de ação exato da
reação de catálise desta protease em particular,
vamos utilizar mecanismos conhecidos de proteases para inferir como a
catálise pela protease de HIV-1 se dá.
Os mecanismos de hidrólise empregados por
proteases em geral envolvem a participação de uma hidroxila da
cadeia lateral de algum aminoácido do sítio
catalítico, como por exemplo, a hidroxila do grupo
carboxílico da cadeia lateral de Asp25.
Veja abaixo a estrutura plana deste aminoácido e a hidroxila
referida.
 |
A
cadeia lateral do aspartato está apresentada em verde. Os oxigênios das hidroxilas do grupo carboxílico da cadeia lateral estão coloridos em vermelho. |
Agora veja
átomo de oxigênio da hidroxila (cadeia lateral) do Asp25
no modelo tridimensional.
| ver oxigênio da hidroxila da cadeia lateral do Asp25 em vermelho |
Uma vez que a
exposição dos aspartatos à cavidade onde o
substrato
se liga é essencial ao funcionamento da enzima, uma
molécula
que ocupe o sítio catalítico irá impedir o bom
funcionamento
da enzima.
Uma
molécula
que liga-se ao sítio ativo, inibindo a enzima é o
inibidor
de protease saquinavir, uma droga anti-HIV.
| mostrar inibidor saquinavir no sítio ativo da protease |
Perceba que o
inibidor ocupa exatamente a cavidade que constitui o sítio ativo
e está em contato com os Asp25.
Veja agora a proteína representada com os
raios atômicos e o inibidor na cavidade.
| ver a proteína
com o inibidor
saquinavir |
Repare que o
inibidor é também simétrico em
relação ao sítio
catalítico.
O inibidor é formado por uma asparagina padrão, uma
fenilalanina
modificada (hidroxi fenilalanina) e por outros grupos, mimetizando em
parte
um peptídeo.