Aula II
Estrutura 1o de proteínas
Ligação peptídica entre aminoácidos
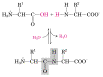
In
vitro, esta reação não acontece pois é muito
devagar/cinéticamente lenta (alta energia de ativação)
e termodinamicamente desfavoravel (DG>0).
In
vivo, está reação pode acontecer: a energia de ativação
é diminuida pela participação de enzimas incluindo
os aminoacil-tRNA sintetases e os ribossomos (com a partcipação
dos RNA mensageiros) e a reação se torna termodinamicamente
favoravel graças ao acoplamento da reação ao hidrólise
de ATP.
A
ligação peptídica pode existir em duas estruturas
de resonância, um com o grupo carbonil com geometria plano trigonal
(sp2) e o nitrogênio com geometria tetragonal (sp3):
O=C-NH(60%
contribuição)
e
o outro com os dois grupos com geometria trigonal e com uma carga negativa
no oxigênio e uma carga positivo no nitrogênio:
-O-C=NH+(40%
contribuição)
Esta
resonância, que implica que a ligação peptídica
tem carater de uma ligação dupla) tem o efeito de deixar
os átomos O=C-N-H num plano.
Para
ficar no mesmo plano, a ligação peptídica pode adoptar
um de dois conformações: trans (180o) ou cis (0o).
A
grande maioria dos ligações peptidicas estão na forma
“trans” (180o) pois nesta conformação, a cadeia
peptídica evita colisões estéricas desfavoráveis
que acontecerão na conformação cis.
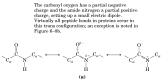
No
caso de uma ligação peptídica que antecede uma prolina,
ambas as conformações tem a mesma probabilidade de acontecer
colisões estéricas desfavoráveis, logo as duas conformações
tem energias semelhantes e as duas formas podem existir. A velocidade de
interconversão entre as conformações cis e trans é
muito lenta.
pI
Ponto
Isoelétrico (pI) é o pH em que um aminoácido, polipetídeo
ou proteína tem uma carga líquida igual a zero (0).
Para
sistemas simples,
o valor do pI pode ser calculado na seguinte forma: a) Identificar as formas
iônicas da molécula que tem cargas líquidas –1, 0 e
+1. b) Identificar o pKa do equilibrio que descreve a interconversão
entre as formas com carga +1 e 0 e
tambem o pKa do equilibrio
que descreve a interconversão entre as formas com carga 0 e –1.
c) Calcular a média dos dois valores do pKa. Esta média é
igual ao pI.
pI
= (pKai + pKaj)/2
Por
exemplo, para glicina que tem pKas de 2,35 e 9,78
pI
= 6,06
Por
exemplo, para ácido aspartico, que tem pKas de 2.0, 3.9 e 9.9
pI
= (2+3,9)/2 = 2,95
Por
exemplo, para o tripeptídeo asp-arg-ala, que tem pKas de 9.7 (alfa-amino),
3.9 (beta-carboxila do asp), 12.5 (guanidino) e 2.4 (carboxi-terminal)
pI
= (9.7+3.9)/2 = 6,8
Conformações permitidas da cadeia principal das proteínas (Diagrama de Ramachandran)
A
conformação da cadeia principal em espaço pode ser
representada pela tabulação de todos os ángulos de
rotoação das suas ligações covalentes.
Na
cadeia principal, tem três ligações covalentes para
cada aminoácido.
Os
ângulos são designadas omega, phi e psi
Um
deles é a ligação peptídica (omega) que é
quase sempre na conformação trans (180o), e as
vezes na conformação cis (0o) nas ligações
peptídicas que antecedam prolinas.
As
ligações phi (entre o grupo NH e carbono alfa) e psi (entre
o carbono alfa e o grupo carbonila) não tem esta restriçào
e tem rotação livre.
MAS:
não todas as combinações phi+psi são permitidas
estericamente – quer dizer: algumas combinações causam colisões
desfavoraveis entre grupos da cadeia principal e/ou as cadeias laterais.
As
combinações permitidas podem ser representadas num diagrama
de Ramachandran.
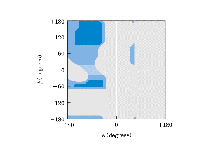
Estruturas
2o de proteínas
Trechos
de aminoácidos consecutivas numa cadeia polipeptídica que
adotam as mesmas conformações nos seus ângulos phi+psi
apresentam estruturas três dimensionais repetitivas na forma de um
hélice ou um zig-zag (que pode ser considerado um hélice
com dois aminoácidos por volta).
Estas
estruturas três-dimiensionais em que aminoácidos consecutivos
adotam conformações (descritas pelos valores dos ângulos
phi e psi) iguais são chamadas estruturas secundárias.
As
estruturas mais comuns são a “alfa hélice” e a“folha
beta”
-revisar
as caraterísticas destas estruturas
Outros
tipos de hélice que diferem no número de aminoácidos
por volta.
Beta-turns
Proteínas fibrosas
“Coiled-coils”
“alfa-queratina”
“Fibroina
de seda”
“Colágeno”
“Elastina”
Estruturas
3o e 4o de proteinas globulares
Interações
importantes para a estabilidade da estrutura das proteínas
Forças
eletrostáticas
Interações
ion-ion
Interações
dipolo-dipolo
Pontes
de hidrogênio
Interações
van der Waals
Efeito
hidrofóbico
Ligações
dissulfeto